
Im letzten Jahrzehnt wurden auf dem Gebiet der molekularen Diagnostik enorme Fortschritte gemacht. Viele neue nukleinsäurebasierte diagnostische Werkzeuge oder Assays wurden entwickelt, die die Analyse von DNA- und RNA-Molekülen in klinischen Proben ermöglichen. Diese Assays werden jetzt routinemäßig zur Überwachung oder zum Nachweis eingesetzt, aber auch um zu entscheiden, welche Therapien bei Patienten am besten wirken würden. Zu diesem Zweck werden spezifische molekulare Sonden und Primer entwickelt. Gensonden werden in verschiedenen Blotting- und In-situ-Hybridisierungsverfahren (ISH) zum Nachweis von Nukleinsäuresequenzen in der Lebensmittelindustrie, im Umweltbereich, in der Medizin und in der Veterinärmedizin eingesetzt, um die Spezifität der Analysen zu verbessern. In der Medizin können sie bei der Identifizierung von Mikroorganismen und der Diagnose von Infektions-, Erb- und anderen Krankheiten helfen. In der Praxis werden doppelte und einfache Standard-DNAs, mRNAs und andere in vitro synthetisierte RNAs als Sonden verwendet. DNA – RNA-Sondentests sind schneller und empfindlicher, so dass viele konventionelle diagnostische Tests für Viren und Bakterien, die eine Kultivierung der Organismen beinhalten, schnell durch molekulare Sondentests ersetzt werden. Während Kulturtests Tage dauern können, können Molekularsonden-Assays innerhalb weniger Stunden oder Minuten durchgeführt werden. Molekulare Sonden können grob in DNA-Sonden und RNA-Sonden kategorisiert werden. cDNA-Sonden und synthetische Oligonukleotid-Sonden können ebenfalls für verschiedene Zwecke eingesetzt werden. Bei Enzo bieten wir ein komplettes Set von Werkzeugen für die Markierung und Detektion von Nukleinsäuren an.
Nukleinsäure-Sonden sind entweder eine einzelsträngige DNA oder eine RNA mit einer starken Affinität zu einer spezifischen DNA- oder RNA-Zielsequenz. Diese Affinität und komplementäre Sequenz ermöglicht die Bindung an spezifische Regionen einer Zielsequenz von Nukleotiden. Der Grad der Homologie zwischen Target und Sonde führt zu einer stabilen Hybridisierung. Bei der Entwicklung einer Sonde muss eine Nukleotidsequenz identifiziert, isoliert, in ausreichender Menge reproduziert und mit einer Markierung versehen werden, die nachgewiesen werden kann. Theoretisch kann jede Nukleinsäure als Sonde verwendet werden, sofern sie markiert werden kann, um die Identifizierung und Quantifizierung der Hybridmoleküle zu ermöglichen, die zwischen der Sonde und der zu identifizierenden Sequenz gebildet werden.
Wahl der Markierung
Sonden können entweder mit radioaktiven Isotopen oder auch mit nicht-radioaktiven Molekülen wie Biotin, Digoxegenin etc. markiert werden. Die Verwendung von radioisotopenmarkierten Sonden ist jedoch durch die kurze Halbwertszeit des Isotops sowie durch wirtschaftliche und ökologische Aspekte der Entsorgung von radioaktivem Abfall begrenzt.
Fortschritte in der Nukleinsäuretechnologie bieten Alternativen zu radioaktiv markierten Sonden. Die Verwendung von nicht-radioaktiven Markern hat mehrere Vorteile wie Sicherheit und höhere Effizienz der Markierungsreaktion. Ein Beispiel ist die Biotin-Markierung von Nukleinsäuren. Bei diesem System wird die Affinität des Glykoproteins Avidin für Biotin ausgenutzt. Diese Sonden können im Voraus als Bulkware hergestellt und bei -20°C für wiederholte Verwendung gelagert werden. Digoxigenin ist eine weitere aus Pflanzen gewonnene Chemikalie, die für die nicht-radioaktive Markierung von Sonden verwendet wird. Ein mit einem Enzym assoziierter Antikörper (Antidigoxigenin – alkalisches Phosphatase-Konjugat) wird für den Nachweis des Vorhandenseins von Digoxigenin verwendet.
Fortschritte in der sequenzspezifischen DNA-Darstellung durch Fluoreszenzmikroskopie wurden mit der Methode der fluoreszierenden Hybridisierung in situ (FISH) erzielt. Diese Art der Markierung ist besonders für die direkte Untersuchung von mikrobiologischen oder zytologischen Proben unter dem Mikroskop geeignet.
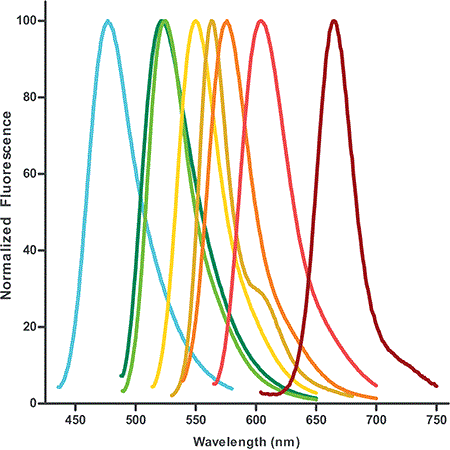
Abbildung 1: Fluoreszenz-Emissionsprofile von verfügbaren fluoreszenzmarkierten dUTPs. Die Farbstoff-DUTPs sind so konzipiert, dass sie sich besonders gut für mehrfarbige Anwendungen eignen, wie z. B. in situ Hybridisierung und Microarray-Analyse.
Was sind DNA-Sonden?
Eine DNA-Sonde ist ein DNA-Fragment, das eine Nukleotidsequenz enthält, die spezifisch für das Gen oder die chromosomale Region von Interesse ist. DNA-Sonden nutzen die Nukleinsäure-Hybridisierung mit spezifisch markierten Sequenzen, um komplementäre Sequenzen in der Testprobe schnell nachzuweisen. Es wurde eine Vielzahl von Methoden zur Markierung von DNA beschrieben. Kurz gesagt, werden diese Methoden verwendet, um endmarkierte oder kontinuierlich markierte Sonden zu erzeugen. Die meisten enzymvermittelten Markierungstechniken sind sehr stark von der Polymeraseaktivität abhängig, die für den Einbau der markierten Nukleotide verantwortlich ist. Darüber hinaus erlaubt die Verwendung von Taq oder anderen thermostabilen DNA-Polymerasen die Durchführung von Markierungsreaktionen bei höheren Temperaturen mittels PCR, wodurch das Auftreten von enzymvermittelten Punktmutationen während der Sondensynthese reduziert wird. Die PCR ist eine exzellente Methode zur Sondensynthese, die sehr geringe Mengen an Templatematerial benötigt. In Gegenwart von entsprechend markierten Nukleotid-Primern werden die PCR-Produkte bereits während der Synthese markiert. Alternativ können die Primer selbst während ihrer eigenen Synthese nicht-isotopisch markiert werden, wodurch die Notwendigkeit der Einbeziehung von markierten Nukleotidvorstufen als Teil der Reaktionsmischung entfällt. Random Priming ist eine Art der Primerverlängerung, bei der eine Mischung aus kleinen Oligonukleotidsequenzen, die als Primer fungieren, an eine hitzedenaturierte doppelsträngige Vorlage anlagern. Die annealisierten Primer werden schließlich Teil der Sonde selbst, da das Klenow-Fragment der DNA-Polymerase I die Primer in 3′-Richtung verlängert und dabei die Markierung einbaut. Die Nick-Translation ist eine der ältesten Sondenmarkierungstechniken. Dabei wird das Rückgrat einer doppelsträngigen DNA mit verdünnten Konzentrationen von DNase I zufällig eingekerbt. Bei extrem niedrigen Konzentrationen kerbt dieses Enzym eine Vorlage an vier oder fünf Stellen ein, wobei eine freie 3′-OH-Gruppe entsteht, die an jeder Einkerbungsstelle als Primer dienen kann. Als nächstes entfernt das Enzym DNA-Polymerase I die nativen Nukleotide von den Sondenmolekülen in 5′→3′-Richtung (Exonuklease-Aktivität), während sie durch ihre 5′→3′-Polymerase-Aktivität durch markierte dNTP-Vorstufen ersetzt werden. Die Nick-Translation ist sowohl für lineare als auch für kovalent geschlossene DNA-Moleküle effizient, und die Markierungsreaktion ist in weniger als einer Stunde abgeschlossen.
Enzo bietet mit dem Nick Translation DNA Labeling System 2.0 eine einfache und effiziente Methode zur Erzeugung von markierter DNA. Das Kit kann eine breite Palette von Fluorophor-markierten, Biotin-markierten und Digoxigenin-markierten Nukleotiden aufnehmen. Zusätzlich zur Wahl der Markierung erlaubt das Design des Kits dem Anwender, die Inkorporation und die Produktgröße zu optimieren, indem das Verhältnis von markiertem dUTP zu dTTP angepasst wird. Der gebrauchsfertige NT-Enzym-Mix ist benutzerfreundlich und minimiert Fehler durch Pipettieren. Durch Nick-Translation markierte Sonden können in vielen verschiedenen Hybridisierungstechniken verwendet werden, darunter: In-situ-Hybridisierung (ISH), Fluoreszenz-in-situ-Hybridisierung (FISH), Screening von Genbanken durch Kolonie- oder Plaque-Hybridisierung, DNA- oder RNA-Transfer-Hybridisierung und kinetische Reassoziationsstudien.
Was sind RNA-Sonden?
RNA-Sonden sind Abschnitte einzelsträngiger RNA, die zum Nachweis des Vorhandenseins von komplementären Nukleinsäuresequenzen (Zielsequenzen) durch Hybridisierung verwendet werden. RNA-Sonden sind in der Regel markiert, z. B. mit Radioisotopen, Epitopen, Biotin oder Fluorophoren, um ihren Nachweis zu ermöglichen. RNA-Sonden als Hybridisierungshilfsmittel sind nach wie vor beliebt, da ihre Verwendung mehrere entscheidende Vorteile bietet. Diese Sonden werden durch in vitro Transkription synthetisiert und können in fast allen Anwendungen DNA-Sonden ersetzen. Hochspezifisch aktive RNA-Sonden oder Ribsonden können auch aus DNA-Vorlagen synthetisiert werden, die in Expressionsvektoren wie SP 6- und T 7-Systemen kloniert sind. RNA-Sonden sind einzelsträngig und bieten mehrere Vorteile gegenüber DNA-Sonden, wie z. B. ein verbessertes Signal oder Hybridisierungsblots. Im Vergleich zu den diversen Methoden zur DNA-Sonden-Synthese gibt es nur eine zuverlässige Methode zur Markierung von RNA-Sonden, nämlich die in vitro Transkription. Aufgrund der intrinsisch labilen Natur von RNA und der Anfälligkeit für RNase-Abbau müssen RNA-Sonden mit der gleichen Sorgfalt behandelt werden wie alle anderen RNA-Präparate.
Die In-vitro-Transkription ist eine zuverlässige und wirtschaftliche Methode zur Herstellung von RNA-Sonden. Durch Transkription einer DNA-Sequenz, die neben einem RNA-Promotor ligiert ist, können große Mengen effizient markierter Sonden mit einheitlicher Länge erzeugt werden. Eine hervorragende Strategie ist es, die zu transkribierende DNA zwischen zwei Promotoren in entgegengesetzter Orientierung zu klonieren. Dadurch kann jeder Strang der klonierten DNA-Sequenz transkribiert werden, um Sense- und Antisense-RNA für Hybridisierungsstudien zu erzeugen. Eine alternative Methode zur Erzeugung kontinuierlich markierter RNA-Sonden durch In-vitro-Transkription ist die Markierung des 5′-Endes des Moleküls. Diese Methode der 5′-Endmarkierung wird umgangssprachlich als Kinasierungsreaktion bezeichnet; sie beinhaltet speziell die Übertragung des γ-Phosphats von ATP auf ein 5′-OH-Substrat der RNA oder DNA (Vorwärtsreaktion). Die Vorwärts-Kinasing-Reaktion ist wesentlich effizienter als die Austauschreaktion, bei der 5′-Phosphate ausgetauscht werden.
Bei der Sondensynthese durch 3′-Endmarkierung werden Nukleotide an das 3′-Ende einer der beiden DNAs angefügt. Die DNA-3′-Endmarkierung wird meist durch eine terminale Transferase katalysiert. Einzel- und doppelsträngige DNA-Moleküle werden durch die Addition von dNTP an 3′-OH-Termini markiert. Auch RNA kann mit Hilfe des Enzyms Poly(A)-Polymerase 3′-endmarkiert werden. Dieses Enzym, das natürlicherweise für die Kernpolyadenylierung vieler heteronukleärer RNAs verantwortlich ist, katalysiert den Einbau von Adenosinmonophosphat. Die Isotopenmarkierung erfordert α-markierte ATP-Vorstufen. Zusätzlich zu ihrem Nutzen in RNA-Sonden-Synthesereaktionen kann die Poly(A)-Polymerase zur Polyadenylierung natürlicher poly(A)-mRNA und anderer RNAs verwendet werden, um die Oligo(dT)-Primer-vermittelte Synthese von cDNA zu unterstützen.
Verwendung von Sonden in Forschungsanwendungen
Beim Northern Blotting wird die zu untersuchende RNA durch Gelelektrophorese fraktioniert. Die Moleküle werden dann auf eine Membran übertragen, die mit der/den markierten Sonde(n) inkubiert wird. Die Hybridisierung der komplementären Sequenzen ermöglicht die Visualisierung der Ziel-RNA-Sequenz. Beim Southern Blotting wird die DNA fraktioniert und auf Membranen übertragen. Die Membranen werden dann mit der/den markierten DNA-Sonde(n) inkubiert. Die Hybridisierung der komplementären Sequenzen ermöglicht die Visualisierung der Ziel-DNA-Sequenz. Andere Anwendungen mit ISH- und FISH-Experimenten ermöglichen die Lokalisierung von RNA- oder DNA-Zielen in Zellen und Geweben. Bei dieser Technik werden kultivierte Zellen oder Gewebeschnittproben für die Hybridisierung und den Nachweis des Gens oder der Zielsequenz von Interesse verwendet. Die Zellen oder Gewebe werden so bearbeitet, dass ihre endogenen Nukleinsäuren an Ort und Stelle fixiert sind, aber für die Hybridisierung mit und den Nachweis durch markierte Sonden zur Verfügung stehen.
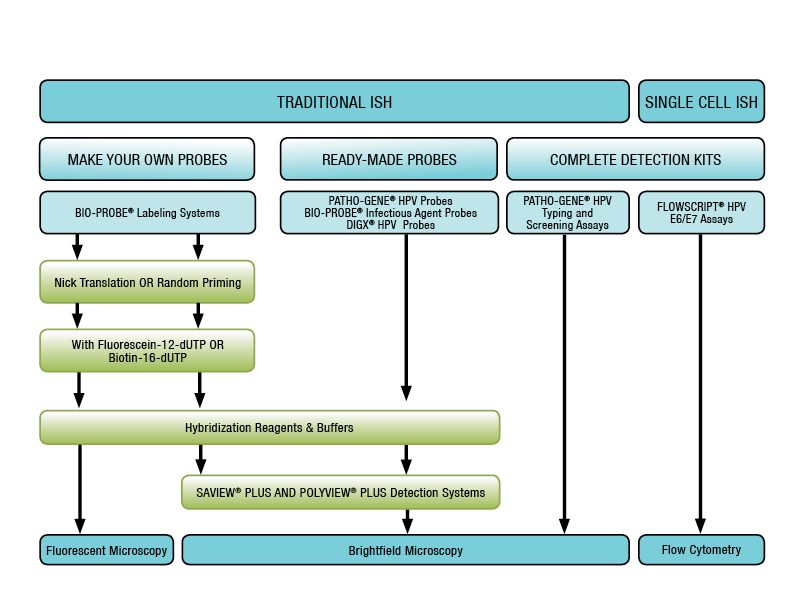
Abbildung 2: Enzo Life Sciences bietet ein komplettes Set von Lösungen für die In-situ-Hybridisierung an, das alles enthält, was Sie für die Markierung, Hybridisierung und Detektion benötigen.
Fortschritte in der Einzelzellanalyse ermöglichen neue Einblicke in phänotypische und funktionelle Heterogenität innerhalb scheinbar identischer Zellpopulationen. Die Techniken zur Erstellung von Profilen und zum Verständnis der RNA-Expression bei Einzelzellauflösung haben in den letzten Jahren rasante Fortschritte gemacht.
Enzo Life Sciences ist ein anerkannter Weltmarktführer in der Bereitstellung von DNA- und RNA-Markierungstechnologien mit mehreren Schlüsselpatenten in der Entwicklung von Biotin- und fluoreszenzmarkierten Nukleotidsonden für Genexpressionsstudien. Wir bieten eine Reihe von Produkten für den Bedarf der Genomforschung an. Für eine einfache und effiziente Methode zur Erzeugung von markierter DNA sehen Sie sich bitte unser Nick-Translations-DNA-Labeling-Kit sowie eine Liste unserer SEEBRIGHT®-Fluoreszenzfarbstoff-dUTPs und unserer Allylamine-dUTP an. Für alle Fragen und Anliegen zu unseren Produkten steht Ihnen unser Technischer Support gerne zur Verfügung.