
En la última década, se han realizado enormes progresos en el campo del diagnóstico molecular. Se han desarrollado muchas herramientas de diagnóstico o ensayos nuevos basados en ácidos nucleicos que permiten el análisis de moléculas de ADN y ARN en muestras clínicas. Estos ensayos se utilizan ahora de forma rutinaria para controlar o detectar, así como para ayudar a decidir qué terapias funcionarían mejor para los pacientes. Para ello se diseñan sondas y cebadores moleculares específicos. Las sondas genéticas se utilizan en varias técnicas de blotting e hibridación in situ (ISH) para la detección de secuencias de ácidos nucleicos en la industria alimentaria, aplicaciones medioambientales, médicas y veterinarias para mejorar la especificidad de los análisis. En medicina, pueden ayudar a la identificación de microorganismos y al diagnóstico de enfermedades infecciosas, hereditarias y de otro tipo. En la práctica, se utilizan como sondas los ADN dobles y simples, los ARNm y otros ARN sintetizados in vitro. Los ensayos con sondas de ADN y ARN son más rápidos y sensibles, por lo que muchas pruebas convencionales de diagnóstico de virus y bacterias que implican el cultivo de los organismos están siendo rápidamente sustituidas por ensayos con sondas moleculares. Mientras que las pruebas de cultivo pueden llevar días, los ensayos con sondas moleculares pueden realizarse en unas pocas horas o minutos. Las sondas moleculares pueden clasificarse en general en sondas de ADN y sondas de ARN; también pueden utilizarse sondas de ADNc y sondas de oligonucleótidos sintéticos para diversos fines. En Enzo, ofrecemos un conjunto completo de herramientas para el etiquetado y la detección de ácidos nucleicos.
Las sondas de ácido nucleico son un ADN monocatenario o un ARN con una fuerte afinidad hacia una secuencia diana específica de ADN o ARN. Esta afinidad y secuencia complementaria permite la unión a regiones específicas de una secuencia de nucleótidos diana. El grado de homología entre la diana y la sonda da lugar a una hibridación estable. Para desarrollar una sonda, hay que identificar una secuencia de nucleótidos, aislarla, reproducirla en cantidad suficiente y marcarla con una etiqueta que pueda ser detectada. En teoría, cualquier ácido nucleico puede utilizarse como sonda siempre que pueda ser etiquetado para permitir la identificación y cuantificación de las moléculas híbridas formadas entre la sonda y la secuencia a identificar.
Elección de la etiqueta
Las sondas pueden ser etiquetadas con isótopos radiactivos o también pueden ser etiquetadas con moléculas no radiactivas como la biotina, digoxegenina, etc. Sin embargo, el uso de sondas marcadas con radioisótopos está limitado por la corta vida media del isótopo y por los aspectos económicos y medioambientales de la eliminación de residuos radiactivos.
Los avances en la tecnología de los ácidos nucleicos ofrecen alternativas a las sondas marcadas radiactivamente. El uso de etiquetas no radiactivas tiene varias ventajas, como la seguridad y la mayor eficiencia de la reacción de etiquetado. Un ejemplo es el etiquetado de ácidos nucleicos con biotina. Este sistema aprovecha la afinidad que la glicoproteína avidina tiene por la biotina. Estas sondas pueden prepararse de antemano a granel y almacenarse a -20°C para usos repetidos. La digoxigenina es otra sustancia química derivada de las plantas que se utiliza para el marcaje no radiactivo de las sondas. Para la detección de la presencia de digoxigenina se utiliza un anticuerpo asociado a una enzima (conjugado antidigoxigenina-fosfatasa alcalina).
Los avances en la obtención de imágenes de ADN específicas de la secuencia mediante microscopía de fluorescencia se han logrado empleando el método de hibridación fluorescente in situ (FISH). Este tipo de etiqueta es especialmente útil para el examen directo de muestras microbiológicas o citológicas al microscopio.
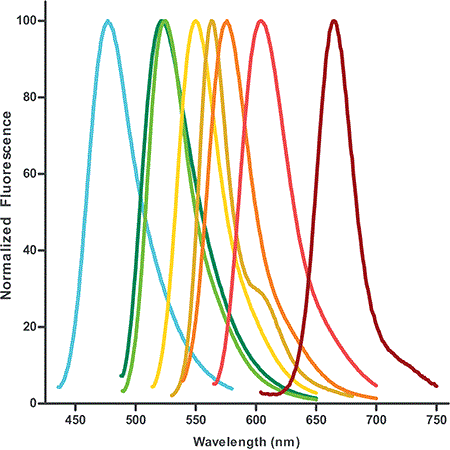
Figura 1: Perfiles de emisión de fluorescencia de los dUTPs marcados con fluorescencia disponibles. Los dUTPs marcados con colorantes están diseñados para funcionar especialmente bien en aplicaciones multicolor, como la hibridación in situ y el análisis de microarrays.
¿Qué son las sondas de ADN?
Una sonda de ADN es un fragmento de ADN que contiene una secuencia de nucleótidos específica para el gen o la región cromosómica de interés. Las sondas de ADN emplean la hibridación del ácido nucleico con secuencias específicamente marcadas para detectar rápidamente secuencias complementarias en la muestra de ensayo. Se han descrito diversas metodologías para etiquetar el ADN. En resumen, estos métodos se utilizan para generar sondas marcadas al final o continuamente. La mayoría de las técnicas de etiquetado mediadas por enzimas dependen en gran medida de la actividad de la polimerasa, que es la responsable de la incorporación de los nucleótidos marcados. Además, el uso de Taq u otras polimerasas de ADN termoestables permite que las reacciones de etiquetado se realicen a temperaturas más altas mediante PCR, reduciendo así la incidencia de mutaciones puntuales mediadas por enzimas durante la síntesis de la sonda. La PCR es un método excelente para la síntesis de sondas, ya que requiere cantidades muy pequeñas de material molde. En presencia de cebadores de nucleótidos debidamente marcados, los productos de la PCR se etiquetan a medida que se sintetizan. Alternativamente, los propios cebadores pueden ser etiquetados de forma no isotópica durante su propia síntesis, negando el requisito de incluir precursores de nucleótidos etiquetados como parte de la mezcla de reacción. El cebado aleatorio es un tipo de extensión de cebadores en el que una mezcla de pequeñas secuencias de oligonucleótidos, que actúan como cebadores, se anuda a una plantilla de doble cadena desnaturalizada por el calor. Los cebadores recocidos acaban formando parte de la propia sonda, ya que el fragmento Klenow de la ADN polimerasa I extiende los cebadores en la dirección 3′ y, al hacerlo, incorpora la etiqueta. La traducción Nick es una de las técnicas más antiguas de etiquetado de sondas. Consiste en mellar aleatoriamente la espina dorsal de un ADN de doble cadena con concentraciones diluidas de DNasa I. A concentraciones extremadamente bajas, esta enzima mella una plantilla en cuatro o cinco sitios, produciendo un grupo 3′-OH libre que puede actuar como cebador en cada lugar de mellado. A continuación, la enzima ADN polimerasa I elimina los nucleótidos nativos de las moléculas de la sonda en la dirección 5′→3′ (actividad exonucleasa) mientras los sustituye por precursores dNTP marcados en virtud de su actividad 5′→3′ polimerasa. La traducción Nick es eficiente tanto para moléculas de ADN lineales como covalentemente cerradas, y la reacción de etiquetado se completa en menos de una hora.
Enzo ofrece un sistema de etiquetado de ADN por traducción Nick 2.0 para proporcionar un método simple y eficiente para generar ADN etiquetado. El kit puede acomodar una amplia gama de nucleótidos marcados con fluoróforos, biotina y digoxigenina. Además de la elección de la etiqueta, el diseño del kit permite al usuario optimizar la incorporación y el tamaño del producto ajustando la proporción de dUTP etiquetado a dTTP. La mezcla de enzimas NT, lista para usar, es fácil de usar y minimiza los errores de pipeteo. Las sondas marcadas por traducción de níquel pueden utilizarse en muchas técnicas de hibridación diferentes, entre ellas: hibridación in situ (ISH), hibridación in situ fluorescente (FISH), cribado de bancos de genes mediante hibridación en colonias o en placas, hibridación por transferencia de ADN o ARN y estudios cinéticos de reasociación.
¿Qué son las sondas de ARN?
Las sondas de ARN son tramos de ARN monocatenario que se utilizan para detectar la presencia de secuencias de ácido nucleico complementarias (secuencias diana) mediante hibridación. Las sondas de ARN suelen estar marcadas, por ejemplo, con radioisótopos, epítopos, biotina o fluoróforos para permitir su detección. Las sondas de ARN como herramientas de hibridación siguen siendo populares debido a varias ventajas clave asociadas a su uso. Estas sondas se sintetizan mediante transcripción in vitro y pueden sustituir a las sondas de ADN en casi todas las aplicaciones. Las sondas de ARN de alta actividad específica o las riboprobes también pueden sintetizarse a partir de plantillas de ADN clonadas en vectores de expresión como los sistemas SP 6 y T 7. Las sondas de ARN son monocatenarias y ofrecen varias ventajas sobre las sondas de ADN, como la mejora de la señal o la hibridación de los blots. En comparación con los diversos métodos de síntesis de sondas de ADN, sólo existe un método fiable para el etiquetado de sondas de ARN, a saber, la transcripción in vitro. Debido a la naturaleza intrínsecamente lábil del ARN y a la susceptibilidad a la degradación de la RNasa, las sondas de ARN deben tratarse con el mismo cuidado que cualquier otra preparación de ARN.
La transcripción in vitro es un método fiable y económico para generar sondas de ARN. Se pueden generar grandes cantidades de sondas eficientemente marcadas de longitud uniforme mediante la transcripción de una secuencia de ADN ligada junto a un promotor de ARN. Una estrategia excelente es clonar el ADN que se va a transcribir entre dos promotores en orientaciones opuestas. Esto permite transcribir cualquiera de las dos cadenas de la secuencia de ADN clonada para generar ARN en sentido y antisentido para los estudios de hibridación. Un método alternativo a la generación de sondas de ARN marcadas de forma continua mediante la transcripción in vitro consiste en marcar el extremo 5′ de la molécula. Este método de etiquetado del extremo 5′ se conoce coloquialmente como reacción de quinasa; implica específicamente la transferencia del γ fosfato del ATP a un sustrato 5′-OH del ARN o del ADN (reacción hacia adelante). La reacción de quinasa hacia adelante es mucho más eficiente que la reacción de intercambio, que implica la sustitución de los 5′ fosfatos.
La síntesis de sondas por etiquetado final 3′ implica la adición de nucleótidos al extremo 3′ de cualquiera de los ADN. El etiquetado del extremo 3′ del ADN suele ser catalizado por la transferasa terminal. Las moléculas de ADN de cadena simple o doble se etiquetan mediante la adición de dNTP a los extremos 3′-OH. El ARN también puede marcarse en su extremo 3′ utilizando la enzima poli(A) polimerasa. Esta enzima, que es la responsable natural de la poliadenilación nuclear de muchos ARN heteronucleares, cataliza la incorporación de monofosfato de adenosina. El etiquetado isotópico requiere precursores de ATP marcados con α. Además de su utilidad en las reacciones de síntesis de sondas de ARN, la poli(A) polimerasa puede utilizarse para poliadenilar naturalmente el ARNm poli(A) y otros ARN con el fin de apoyar la síntesis de ADNc mediada por cebadores oligo(dT).
Uso de las sondas en aplicaciones de investigación
En el Northern blotting, el ARN en estudio se fracciona mediante electroforesis en gel. A continuación, las moléculas se transfieren a una membrana que se incuba con la(s) sonda(s) marcada(s). La hibridación de las secuencias complementarias permite visualizar la secuencia del ARN objetivo. La técnica de Southern blot implica el fraccionamiento y la transferencia del ADN a las membranas. A continuación, las membranas se incuban con la(s) sonda(s) de ADN marcada(s). La hibridación de las secuencias complementarias permite visualizar la secuencia de ADN objetivo. Otras aplicaciones que implican experimentos ISH y FISH permiten la localización de objetivos de ARN o ADN en células y tejidos. Esta técnica utiliza células cultivadas o muestras de secciones de tejido para la hibridación y detección del gen o secuencia diana de interés. Las células o los tejidos se procesan para que sus ácidos nucleicos endógenos se fijen en su lugar, pero estén disponibles para la hibridación y la detección mediante sondas marcadas.
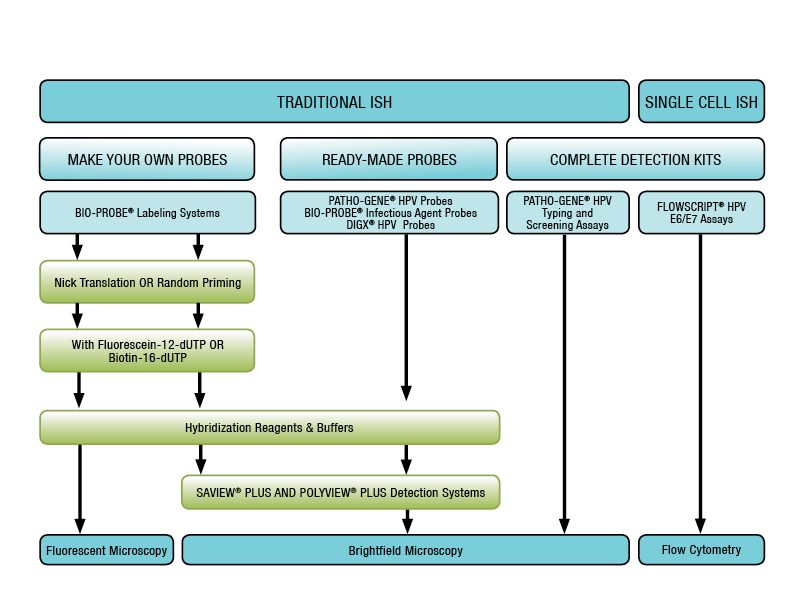
Figura 2: Enzo Life Sciences ofrece un conjunto completo de soluciones para la hibridación in situ, proporcionando todo lo necesario para el etiquetado, hibridación y detección.
Los avances en las tecnologías de análisis unicelular están proporcionando nuevos conocimientos sobre la heterogeneidad fenotípica y funcional dentro de poblaciones celulares aparentemente idénticas. Las técnicas para perfilar y comprender la expresión del ARN con una resolución unicelular han progresado rápidamente en los últimos años.
Enzo Life Sciences es un líder mundial reconocido en el suministro de tecnologías de etiquetado de ADN y ARN con varias patentes clave en el desarrollo de sondas de nucleótidos etiquetados con biotina y fluorescentes para estudios de expresión génica. Ofrecemos una gama de productos para las necesidades de la investigación genómica. Si desea un método sencillo y eficaz para generar ADN etiquetado, consulte nuestro kit de etiquetado de ADN de traducción Nick, así como una lista de nuestros SEEBRIGHT® dUTPs de colorante fluorescente y nuestro Allylamine-dUTP. Para todas las preguntas e inquietudes relacionadas con cualquiera de nuestros productos, nuestro equipo de soporte técnico está aquí para ayudar.