
Na última década, foram feitos enormes progressos no campo do diagnóstico molecular. Foram desenvolvidas muitas novas ferramentas ou ensaios de diagnóstico baseados em ácido nucleico que permitem a análise de moléculas de ADN e RNA em amostras clínicas. Estes ensaios são agora utilizados rotineiramente para monitorização ou detecção, bem como para ajudar a decidir quais as terapias que funcionariam melhor para os pacientes. Sondas moleculares e primários específicos são concebidos para este fim. As sondas genéticas são utilizadas em várias técnicas de blotting e hibridação in situ (ISH) para a detecção de sequências de ácidos nucleicos na indústria alimentar, aplicações ambientais, médicas e veterinárias para melhorar a especificidade das análises. Na medicina, podem ajudar na identificação de microrganismos e no diagnóstico de doenças infecciosas, hereditárias, e outras. Na prática, os DNAs de padrão duplo e único, mRNAs, e outros RNAs sintetizados in vitro são todos utilizados como sondas. Os ensaios com sondas de ADN – RNA são mais rápidos e sensíveis, de modo que muitos testes de diagnóstico convencionais para vírus e bactérias envolvendo a cultura dos organismos estão a ser rapidamente substituídos por ensaios de sondas moleculares. Enquanto os testes de cultura podem demorar dias, os ensaios de sondas moleculares podem ser realizados em poucas horas ou minutos. As sondas moleculares podem ser amplamente categorizadas em sondas de ADN e sondas de RNA, sondas de cDNA e sondas de oligonucleótidos sintéticos também podem ser utilizadas para vários fins. Na Enzo, oferecemos um conjunto completo de ferramentas para rotulagem e detecção de ácido nucleico.
Sondas de ácido nucleico são ou um único ADN encalhado ou um RNA com uma forte afinidade com uma sequência alvo específica de ADN ou RNA. Esta afinidade e sequência complementar permite a ligação a regiões específicas de uma sequência alvo de nucleótidos. O grau de homologia entre o alvo e a sonda resulta numa hibridação estável. Ao desenvolver uma sonda, uma sequência de nucleótidos deve ser identificada, isolada, reproduzida em quantidade suficiente, e marcada com uma etiqueta que possa ser detectada. Em teoria, qualquer ácido nucleico pode ser utilizado como uma sonda desde que possa ser rotulado para permitir a identificação e quantificação das moléculas híbridas formadas entre a sonda e a sequência a ser identificada.
h2>Escolha de EtiquetaAs sondas podem ser etiquetadas quer por isótopos radioactivos quer também podem ser etiquetadas com moléculas não radioactivas tais como biotina, digoxegenina, etc. Contudo, a utilização de sondas rotuladas com radioisótopos é limitada pela curta semi-vida do isótopo, e pelos aspectos económicos e ambientais da eliminação de resíduos radioactivos.
Os avanços na tecnologia de ácido nucleico oferecem alternativas às sondas rotuladas radioactivamente. A utilização de etiquetas não radioactivas tem várias vantagens, tais como segurança e maior eficiência da reacção de etiquetagem. Um exemplo é a etiquetagem biotina dos ácidos nucleicos. Este sistema explora a afinidade que a glicoproteína avidina tem com a biotina. Estas sondas podem ser preparadas com antecedência a granel e armazenadas a -20°C para usos repetidos. A digoxigenina é outro químico derivado de plantas e utilizado para a etiquetagem não radioactiva das sondas. Um anticorpo associado a uma enzima (antidigoxigenina – conjugado de fosfatase alcalina) é utilizado para a detecção da presença de digoxigenina.
Progresso na imagem de ADN específico da sequência por microscopia de fluorescência foi alcançado através do método de hibridação fluorescente in situ (FISH). Este tipo de rótulo é especialmente útil para o exame directo de amostras microbiológicas ou citológicas sob o microscópio.
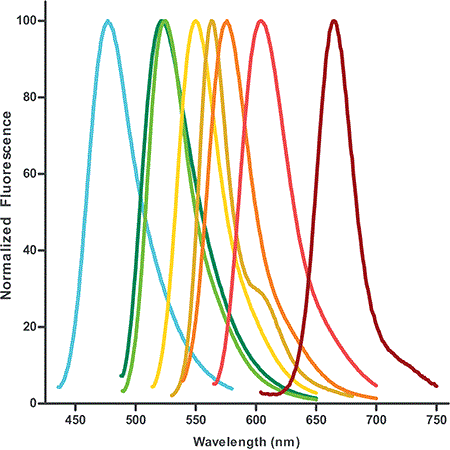 br>Figure 1: Perfis de emissão de fluorescência dos dUTPs com rótulos fluorescentes disponíveis. Os dUTPs tingidos são concebidos para ter um desempenho especialmente bom em aplicações multicoloridas, tais como hibridação in situ e análise de microarranjos.
br>Figure 1: Perfis de emissão de fluorescência dos dUTPs com rótulos fluorescentes disponíveis. Os dUTPs tingidos são concebidos para ter um desempenho especialmente bom em aplicações multicoloridas, tais como hibridação in situ e análise de microarranjos.
h2>O que são sondas de ADN?Uma sonda de ADN é um fragmento de ADN que contém uma sequência nucleotídica específica para o gene ou região cromossómica de interesse. As sondas de ADN empregam a hibridação do ácido nucleico com sequências especificamente rotuladas para detectar rapidamente sequências complementares na amostra de teste. Foi descrita uma variedade de metodologias para etiquetar o ADN. Em suma, estes métodos são utilizados para gerar sondas rotuladas ou continuamente rotuladas. A maioria das técnicas de rotulagem mediadas por enzimas dependem muito da actividade da polimerase, que é responsável pela incorporação dos nucleótidos rotulados. Além disso, a utilização de Taq ou outras polimerases de ADN termoestáveis permite a realização de reacções de rotulagem a temperaturas mais elevadas via PCR, reduzindo assim a incidência de mutações pontuais mediadas por enzimas durante a síntese das sondas. A PCR é um excelente método para síntese de sondas, exigindo quantidades muito pequenas de material de modelo. Na presença de primários de nucleótidos devidamente rotulados, os produtos PCR são rotulados à medida que estão a ser sintetizados. Em alternativa, os próprios iniciadores podem ser rotulados não-isotopicamente durante a sua própria síntese, negando a exigência da inclusão de precursores de nucleótidos rotulados como parte da mistura de reacção. O primer aleatório é um tipo de extensão do primer em que uma mistura de pequenas sequências de oligonucleótidos, actuando como primers, recolem a um modelo de dupla cadeia desnaturada por calor. Os primários recozidos acabam por fazer parte da própria sonda, porque o fragmento de Klenow de DNA polimerase I estende os primários na direcção 3′ e, ao fazê-lo, incorpora a etiqueta. A tradução Nick é uma das mais antigas técnicas de etiquetagem de sondas. Envolve o corte aleatório da espinha dorsal de um ADN de cadeia dupla com concentrações diluídas de DNase I. Em concentrações extremamente baixas, esta enzima corta um modelo em quatro ou cinco locais, produzindo um grupo 3′-OH gratuito que pode actuar como primário em cada local de corte. Em seguida, a enzima DNA polimerase I remove os nucleótidos nativos das moléculas da sonda na direcção 5′→3′ (actividade de exonuclease), substituindo-os por precursores dNTP rotulados em virtude da sua actividade de polimerase 5′→3′. A tradução Nick é eficiente tanto para moléculas de ADN lineares como covalentemente fechadas, e a reacção de etiquetagem é completada em menos de uma hora.
Enzo oferece um Sistema de Etiquetagem de ADN Nick Translation 2.0 para fornecer um método simples e eficiente de geração de ADN rotulado. O kit pode acomodar uma vasta gama de nucleótidos marcados com fluoróforos, marcados com biotina, e marcados com digoxigenina. Além da escolha da etiqueta, o desenho do kit permite ao utilizador optimizar a incorporação e o tamanho do produto, ajustando a relação entre o rotulado e o dTTP. A mistura enzimática NT pronta a usar é de fácil utilização e minimiza os erros de pipetagem. As sondas rotuladas por tradução de nick podem ser utilizadas em muitas técnicas diferentes de hibridação, incluindo: hibridação in situ (ISH), hibridação fluorescente in situ (FISH), bancos de genes de rastreio por hibridação de colónias ou placas, hibridação de transferência de ADN ou RNA, e estudos cinéticos de re-associação.
O que são sondas de RNA?
Sondas de RNA são estiramentos de RNA de cadeia única utilizados para detectar a presença de sequências de ácidos nucleicos complementares (sequências alvo) por hibridação. As sondas de RNA são normalmente etiquetadas, por exemplo com radioisótopos, epitopos, biotina ou fluoróforos para permitir a sua detecção. As sondas de ARN como ferramentas de hibridação continuam a ser populares devido a várias vantagens chave associadas à sua utilização. Estas sondas são sintetizadas por transcrição in vitro e podem ser substituídas por sondas de ADN em quase todas as aplicações. As sondas de ARN de alta actividade específica ou as sondas de riboprobes também podem ser sintetizadas a partir de modelos de ADN clonados em vectores de expressão, tais como os sistemas SP 6 e T 7. As sondas de RNA são de fio único e oferecem várias vantagens em relação às sondas de ADN, incluindo o melhoramento do sinal ou das manchas de hibridação. Em comparação com os diversos métodos de síntese de sondas de ADN, existe apenas um método fiável para a etiquetagem de sondas de ARN, nomeadamente a transcrição in vitro. Devido à natureza intrinsecamente labial do RNA e à susceptibilidade à degradação do RNase, as sondas de RNA devem ser tratadas com o mesmo cuidado que quaisquer outras preparações de RNA.
transcrição in vitro é um método fiável e económico para gerar sondas de RNA. Grandes quantidades de sondas eficazmente rotuladas de comprimento uniforme podem ser geradas por transcrição de uma sequência de ADN ligada ao lado de um promotor de RNA. Uma excelente estratégia é clonar o ADN a ser transcrito entre dois promotores em orientações opostas. Isto permite que qualquer uma das vertentes da sequência de ADN clonado seja transcrita de modo a gerar RNA sensorial e antisense para estudos de hibridação. Um método alternativo para gerar sondas de RNA continuamente rotuladas por transcrição in vitro é rotular a extremidade da molécula 5′. Este método de etiquetagem final de 5′ é coloquialmente conhecido como reacção de kinasing; envolve especificamente a transferência do fosfato de ATP γ para um substrato de RNA ou ADN 5′-OH (reacção de avanço). A reacção de kinasing frontal é muito mais eficiente do que a reacção de troca que envolve a substituição de 5′ fosfatos.
Síntese da sonda por 3′ end-labeling envolve a adição de nucleótidos ao 3′ end-labeling de qualquer ADN. A etiquetagem final do DNA 3′ é mais frequentemente catalisada pela transferase terminal. As moléculas de ADN de cadeia simples e dupla são rotuladas pela adição de dNTP ao terminal 3′-OH. O RNA também pode ser etiquetado em 3′ usando a enzima poli(A) polimerase. Esta enzima, que é naturalmente responsável pela poliadenilação nuclear de muitos RNAs heteronucleares, catalisa a incorporação de Adenosina Mono Fosfato. A etiquetagem isotópica requer precursores ATP marcados com α. Para além da sua utilidade nas reacções de síntese de RNA, a poli(A) polimerase pode ser utilizada para poliadenilato naturalmente poli(A)- mRNA e outros RNAs, a fim de apoiar a síntese de cDNA mediada por primer oligo(dT).
h2> Utilização de Sondas em Aplicações de Investigação Em Northern blotting, o RNA em estudo é fraccionado por electroforese em gel. As moléculas são então transferidas para uma membrana que é incubada com a(s) sonda(s) rotulada(s). A hibridação de sequências complementares permite a visualização da sequência de RNA alvo. A mancha sul envolve o fraccionamento e a transferência do ADN para as membranas. As membranas são então incubadas com a(s) sonda(s) de ADN rotulada(s). A hibridação de sequências complementares permite a visualização da sequência de ADN alvo. Outras aplicações envolvendo experiências ISH e FISH permitem a localização de RNA ou alvos de ADN em células e tecidos. Esta técnica utiliza células cultivadas ou amostras de secção de tecidos para hibridização e detecção do gene ou da sequência alvo de interesse. As células ou tecidos são processados para que os seus ácidos nucleicos endógenos sejam fixados no local, mas disponíveis para hibridização e detecção por sondas rotuladas.
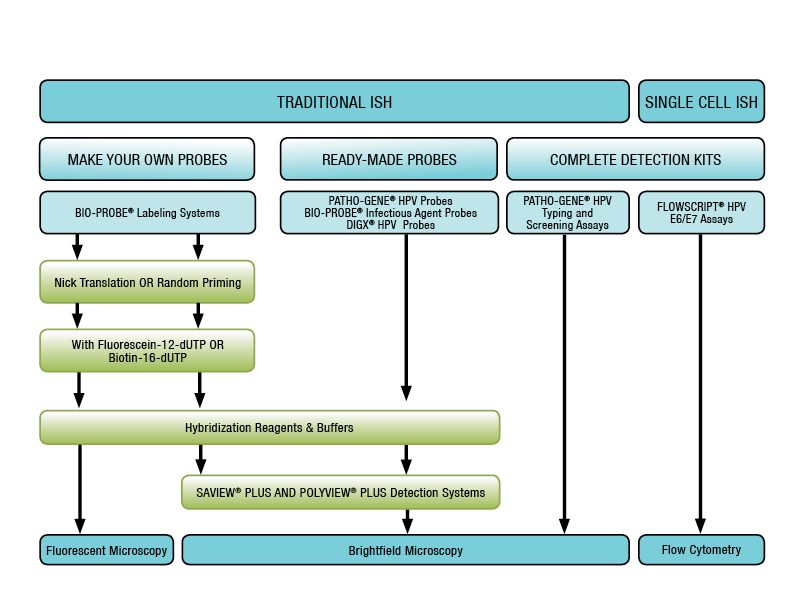 br>Figure 2: Enzo Life Sciences oferece um conjunto completo de soluções para hibridação in situ, fornecendo tudo o que é necessário para a etiquetagem, hibridação e detecção. >br>
br>Figure 2: Enzo Life Sciences oferece um conjunto completo de soluções para hibridação in situ, fornecendo tudo o que é necessário para a etiquetagem, hibridação e detecção. >br>br>Avanços nas tecnologias de análise de célula única estão a fornecer novos conhecimentos sobre a heterogeneidade fenotípica e funcional dentro de populações de células aparentemente idênticas. As técnicas de caracterização e compreensão da expressão do ARN na resolução de célula única têm progredido rapidamente nos últimos anos.
Enzo Life Sciences é um líder mundial reconhecido no fornecimento de tecnologias de etiquetagem de ADN e RNA com várias patentes chave no desenvolvimento de biotina e sonda de nucleótidos com rótulo fluorescente para estudos de expressão genética. Oferecemos uma gama de produtos para necessidades de investigação genómica. Para um método simples e eficiente de geração de ADN rotulado, consulte o nosso kit de etiquetagem de ADN de tradução Nick, bem como uma lista dos nossos corantes fluorescentes SEEBRIGHT®dUTP e o nosso Allylamine-dUTP. Para todas as questões e preocupações relativas a qualquer um dos nossos produtos, a nossa Equipa de Apoio Técnico está aqui para ajudar.