
Nell’ultimo decennio, sono stati fatti enormi progressi nel campo della diagnostica molecolare. Sono stati sviluppati molti nuovi strumenti diagnostici o saggi basati sugli acidi nucleici che permettono di analizzare le molecole di DNA e RNA nei campioni clinici. Questi test sono ora utilizzati di routine per il monitoraggio o la rilevazione, così come per aiutare a decidere quali terapie potrebbero funzionare meglio per i pazienti. Sonde molecolari e primer specifici sono progettati per questo scopo. Le sonde geniche sono utilizzate in varie tecniche di blotting e ibridazione in situ (ISH) per il rilevamento di sequenze di acidi nucleici nell’industria alimentare, ambientale, medica e veterinaria per migliorare la specificità delle analisi. In medicina, possono aiutare nell’identificazione di microrganismi e nella diagnosi di malattie infettive, ereditarie e di altro tipo. In pratica, come sonde vengono utilizzati DNA standard doppi e singoli, mRNA e altri RNA sintetizzati in vitro. I test con sonde a DNA e RNA sono più veloci e sensibili, così che molti test diagnostici convenzionali per virus e batteri che coinvolgono la coltura degli organismi sono stati rapidamente sostituiti da test con sonde molecolari. Mentre i test di coltura possono richiedere giorni, i test con sonda molecolare possono essere eseguiti in poche ore o minuti. Le sonde molecolari possono essere ampiamente classificate in sonde a DNA e sonde a RNA, anche le sonde a cDNA e le sonde oligonucleotidiche sintetiche possono essere utilizzate per vari scopi. Noi di Enzo offriamo una serie completa di strumenti per l’etichettatura e la rilevazione degli acidi nucleici.
Le sonde di acido nucleico sono un DNA a singolo filamento o un RNA con una forte affinità verso una specifica sequenza target di DNA o RNA. Questa affinità e la sequenza complementare permettono di legarsi a regioni specifiche di una sequenza bersaglio di nucleotidi. Il grado di omologia tra bersaglio e sonda determina un’ibridazione stabile. Nello sviluppo di una sonda, una sequenza di nucleotidi deve essere identificata, isolata, riprodotta in quantità sufficiente e contrassegnata con un’etichetta che possa essere rilevata. In teoria, qualsiasi acido nucleico può essere usato come sonda, purché possa essere etichettato per permettere l’identificazione e la quantificazione delle molecole ibride formate tra la sonda e la sequenza da identificare.
Scelta dell’etichetta
Le sonde possono essere etichettate con isotopi radioattivi o anche con molecole non radioattive come la biotina, la digossegenina ecc. Tuttavia, l’uso di sonde marcate con radioisotopi è limitato dalla breve emivita dell’isotopo e dagli aspetti economici e ambientali dello smaltimento dei rifiuti radioattivi.
I progressi nella tecnologia degli acidi nucleici offrono alternative alle sonde marcate radioattivamente. L’uso di etichette non radioattive ha diversi vantaggi come la sicurezza e la maggiore efficienza della reazione di etichettatura. Un esempio è l’etichettatura con biotina degli acidi nucleici. Questo sistema sfrutta l’affinità che la glicoproteina avidina ha per la biotina. Queste sonde possono essere preparate in anticipo alla rinfusa e conservate a -20°C per usi ripetuti. La digossigenina è un’altra sostanza chimica derivata dalle piante e utilizzata per l’etichettatura non radioattiva delle sonde. Un anticorpo associato a un enzima (coniugato antidigoxigenina – fosfatasi alcalina) viene utilizzato per la rilevazione della presenza di digossigenina.
I progressi nell’imaging del DNA sequenza-specifico tramite microscopia a fluorescenza sono stati raggiunti impiegando il metodo dell’ibridazione fluorescente in situ (FISH). Questo tipo di etichetta è particolarmente utile per l’esame diretto di campioni microbiologici o citologici al microscopio.
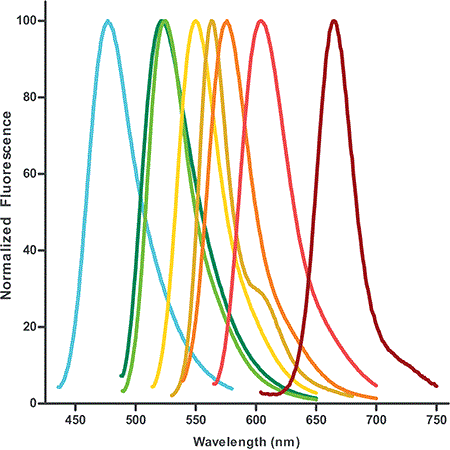
Figura 1: Profili di emissione di fluorescenza dei dUTP marcati con fluorescenza disponibili. I dye-dUTP sono progettati per funzionare particolarmente bene in applicazioni multicolore, come l’ibridazione in situ e l’analisi microarray.
Cosa sono le sonde a DNA?
Una sonda a DNA è un frammento di DNA che contiene una sequenza nucleotidica specifica per il gene o la regione cromosomica di interesse. Le sonde a DNA impiegano l’ibridazione dell’acido nucleico con sequenze specificamente marcate per rilevare rapidamente le sequenze complementari nel campione in esame. Sono state descritte diverse metodologie per etichettare il DNA. In breve, questi metodi sono usati per generare sonde etichettate all’estremità o in modo continuo. La maggior parte delle tecniche di etichettatura mediate da enzimi sono molto dipendenti dall’attività della polimerasi, che è responsabile dell’incorporazione dei nucleotidi etichettati. Inoltre, l’uso di Taq o di altre DNA polimerasi termostabili permette di eseguire reazioni di etichettatura a temperature più elevate tramite PCR, riducendo così l’incidenza di mutazioni puntiformi mediate dall’enzima durante la sintesi della sonda. La PCR è un metodo eccellente per la sintesi della sonda, che richiede quantità molto piccole di materiale campione. In presenza di primer nucleotidici opportunamente etichettati, i prodotti della PCR sono etichettati mentre vengono sintetizzati. In alternativa, i primer stessi possono essere etichettati non isotopicamente durante la loro stessa sintesi, negando la necessità di includere precursori nucleotidici etichettati come parte della miscela di reazione. Il priming casuale è un tipo di estensione del primer in cui una miscela di piccole sequenze oligonucleotidiche, che fungono da primer, si annebbia su un template a doppio filamento denaturato al calore. I primer annebbiati alla fine diventano parte della sonda stessa, perché il frammento Klenow della DNA polimerasi I estende i primer nella direzione 3′ e, così facendo, incorpora l’etichetta. La traduzione Nick è una delle più antiche tecniche di etichettatura della sonda. Si tratta di intaccare in modo casuale la spina dorsale di un DNA a doppio filamento con concentrazioni diluite di DNasi I. A concentrazioni estremamente basse, questo enzima intacca un modello in quattro o cinque siti, producendo un gruppo 3′-OH libero che può agire come un primer in ogni posizione di intaccatura. Successivamente, l’enzima DNA polimerasi I rimuove i nucleotidi nativi dalle molecole della sonda in direzione 5′→3′ (attività esonucleasica) mentre li sostituisce con precursori dNTP marcati in virtù della sua attività 5′→3′ polimerasi. La traduzione Nick è efficiente sia per le molecole di DNA lineari che per quelle covalentemente chiuse, e le reazioni di etichettatura sono completate in meno di un’ora.
Enzo offre un Nick Translation DNA Labeling System 2.0 per fornire un metodo semplice ed efficiente per generare DNA etichettato. Il kit può ospitare una vasta gamma di nucleotidi marcati con fluorofori, biotina e digossigenina. Oltre alla scelta dell’etichetta, il design del kit permette all’utente di ottimizzare l’incorporazione e la dimensione del prodotto regolando il rapporto tra dUTP etichettato e dTTP. La miscela enzimatica NT pronta all’uso è facile da usare e riduce al minimo gli errori di pipettaggio. Le sonde marcate con nick translation possono essere utilizzate in diverse tecniche di ibridazione, tra cui: ibridazione in situ (ISH), ibridazione fluorescente in situ (FISH), screening di banche di geni tramite ibridazione di colonie o placche, ibridazione di trasferimento di DNA o RNA e studi di cinetica di riassociazione.
Cosa sono le sonde RNA?
Le sonde RNA sono tratti di RNA a singolo filamento utilizzati per rilevare la presenza di sequenze di acido nucleico complementari (sequenze target) tramite ibridazione. Le sonde a RNA sono solitamente marcate, per esempio con radioisotopi, epitopi, biotina o fluorofori per permettere la loro rilevazione. Le sonde a RNA come strumenti di ibridazione rimangono popolari a causa di diversi vantaggi chiave associati al loro utilizzo. Queste sonde sono sintetizzate dalla trascrizione in vitro e possono essere sostituite dalle sonde a DNA in quasi tutte le applicazioni. Sonde RNA ad alta attività specifica o riboprobe possono anche essere sintetizzate da modelli di DNA clonati in vettori di espressione come i sistemi SP 6 e T 7. Le sonde a RNA sono a singolo filamento e offrono diversi vantaggi rispetto alle sonde a DNA, tra cui un segnale migliore o blot di ibridazione. Rispetto ai diversi metodi per la sintesi di sonde di DNA, c’è solo un metodo affidabile per etichettare le sonde di RNA, cioè la trascrizione in vitro. A causa della natura intrinsecamente labile dell’RNA e della suscettibilità alla degradazione della RNasi, le sonde RNA devono essere trattate con la stessa cura di qualsiasi altra preparazione di RNA. a trascrizione in vitro è un metodo affidabile ed economico per la generazione di sonde RNA. Grandi quantità di sonde efficientemente etichettate di lunghezza uniforme possono essere generate dalla trascrizione di una sequenza di DNA legata accanto a un promotore di RNA. Una strategia eccellente è quella di clonare il DNA da trascrivere tra due promotori in orientamenti opposti. Questo permette di trascrivere entrambi i filamenti della sequenza di DNA clonata per generare RNA senso e antisenso per gli studi di ibridazione. Un metodo alternativo alla generazione di sonde di RNA marcate in modo continuo mediante trascrizione in vitro è quello di marcare l’estremità 5′ della molecola. Questo metodo di etichettatura dell’estremità 5′ è noto colloquialmente come la reazione di kinasing; in particolare comporta il trasferimento del fosfato γ dell’ATP a un substrato 5′-OH di RNA o DNA (reazione forward). La reazione di kinasing in avanti è molto più efficiente della reazione di scambio che comporta la sostituzione di 5′ fosfati.
La sintesi della sonda tramite 3′ end-labeling comporta l’aggiunta di nucleotidi all’estremità 3′ del DNA. L’etichettatura dell’estremità 3′ del DNA è più spesso catalizzata dalla transferasi terminale. Le molecole di DNA a singolo e doppio filamento vengono etichettate mediante l’aggiunta di dNTP ai termini 3′-OH. Anche l’RNA può essere etichettato in 3′ usando l’enzima polimerasi (A). Questo enzima, che è naturalmente responsabile della poliadenilazione nucleare di molti RNA eteronucleari, catalizza l’incorporazione di adenosina monofosfato. L’etichettatura isotopica richiede precursori di ATP marcati α. Oltre alla sua utilità nelle reazioni di sintesi della sonda RNA, la polimerasi poly(A) può essere usata per poliadenilare naturalmente l’mRNA poly(A)- e altri RNA per supportare la sintesi mediata da primer oligo(dT) di cDNA.
Uso delle sonde nelle applicazioni di ricerca
Nel Northern blotting, l’RNA in studio viene frazionato mediante elettroforesi su gel. Le molecole vengono poi trasferite su una membrana che viene incubata con le sonde marcate. L’ibridazione delle sequenze complementari permette la visualizzazione della sequenza di RNA target. Il Southern blotting comporta il frazionamento e il trasferimento del DNA su membrane. Le membrane vengono poi incubate con le sonde di DNA marcate. L’ibridazione delle sequenze complementari permette la visualizzazione della sequenza di DNA target. Altre applicazioni che coinvolgono esperimenti ISH e FISH permettono la localizzazione di RNA o DNA target in cellule e tessuti. Questa tecnica utilizza cellule coltivate o campioni di sezioni di tessuto per l’ibridazione e il rilevamento del gene o della sequenza target di interesse. Le cellule o i tessuti sono trattati in modo che i loro acidi nucleici endogeni siano fissati in posizione, ma disponibili per l’ibridazione e il rilevamento da parte di sonde marcate.
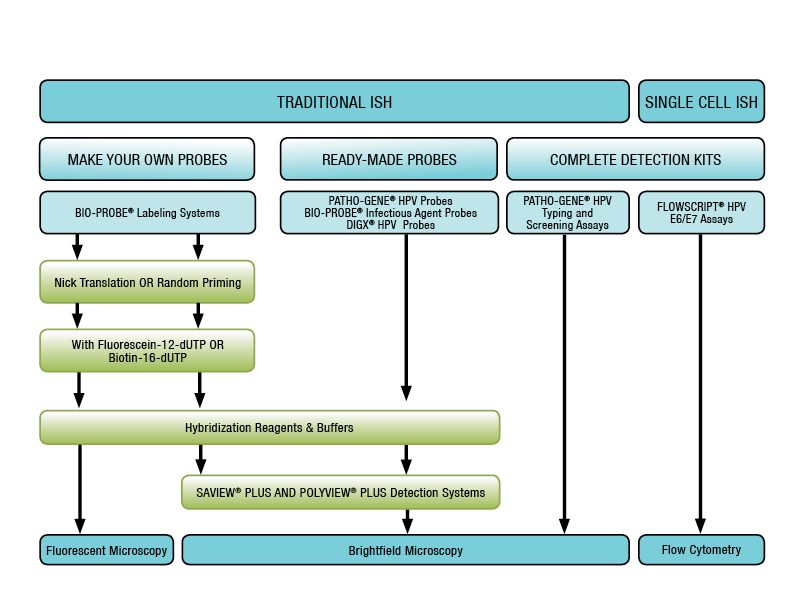
Figura 2: Enzo Life Sciences offre una serie completa di soluzioni per l’ibridazione in situ, fornendo tutto il necessario per l’etichettatura, l’ibridazione e la rilevazione.
I progressi nelle tecnologie di analisi delle singole cellule stanno fornendo nuove conoscenze sull’eterogeneità fenotipica e funzionale all’interno di popolazioni di cellule apparentemente identiche. Le tecniche per il profiling e la comprensione dell’espressione dell’RNA alla risoluzione della singola cellula sono progredite rapidamente negli ultimi anni.
Enzo Life Sciences è un leader globale riconosciuto nella fornitura di tecnologie di etichettatura del DNA e dell’RNA con diversi brevetti chiave nello sviluppo di sonde nucleotidiche etichettate con biotina e fluorescenza per studi di espressione genica. Offriamo una gamma di prodotti per le esigenze della ricerca genomica. Per un metodo semplice ed efficiente per la generazione di DNA etichettato, si prega di controllare il nostro kit di etichettatura del DNA per la traduzione di Nick, nonché un elenco dei nostri SEEBRIGHT® dye-dUTP fluorescenti e il nostro Allylamine-dUTP. Per tutte le domande e i dubbi riguardanti uno qualsiasi dei nostri prodotti, il nostro team di supporto tecnico è qui per assistere.