
Au cours de la dernière décennie, d’énormes progrès ont été réalisés dans le domaine du diagnostic moléculaire. De nombreux nouveaux outils de diagnostic ou tests basés sur les acides nucléiques ont été développés pour permettre l’analyse des molécules d’ADN et d’ARN dans les échantillons cliniques. Ces tests sont désormais utilisés en routine pour la surveillance ou la détection, ainsi que pour aider à déterminer les thérapies les plus efficaces pour les patients. Des sondes moléculaires et des amorces spécifiques sont conçues à cet effet. Les sondes génétiques sont utilisées dans diverses techniques de blotting et d’hybridation in situ (ISH) pour la détection de séquences d’acides nucléiques dans l’industrie alimentaire, l’environnement, les applications médicales et vétérinaires, afin d’améliorer la spécificité des analyses. En médecine, ils peuvent aider à l’identification des micro-organismes et au diagnostic des maladies infectieuses, héréditaires et autres. Dans la pratique, on utilise comme sondes des ADN, des ARNm et d’autres ARN double et simple standard synthétisés in vitro. Les tests par sonde ADN-ARN sont plus rapides et plus sensibles, de sorte que de nombreux tests de diagnostic classiques pour les virus et les bactéries, qui impliquent la culture des organismes, sont rapidement remplacés par des tests par sonde moléculaire. Alors que les tests de culture peuvent prendre plusieurs jours, les tests par sonde moléculaire peuvent être réalisés en quelques heures ou minutes. Les sondes moléculaires peuvent être largement catégorisées en sondes ADN et sondes ARN, les sondes ADNc et les sondes oligonucléotides synthétiques peuvent également être utilisées à des fins diverses. Chez Enzo, nous offrons un ensemble complet d’outils pour le marquage et la détection d’acide nucléique.
Les sondes d’acide nucléique sont soit un ADN simple brin ou un ARN avec une forte affinité vers une séquence cible spécifique d’ADN ou d’ARN. Cette affinité et la séquence complémentaire permettent la liaison à des régions spécifiques d’une séquence cible de nucléotides. Le degré d’homologie entre la cible et la sonde entraîne une hybridation stable. Lors de la mise au point d’une sonde, une séquence de nucléotides doit être identifiée, isolée, reproduite en quantité suffisante et marquée avec un marqueur qui peut être détecté. En théorie, tout acide nucléique peut être utilisé comme sonde à condition qu’il puisse être marqué pour permettre l’identification et la quantification des molécules hybrides formées entre la sonde et la séquence à identifier.
Choix du marquage
Les sondes peuvent être marquées soit par des isotopes radioactifs ou peuvent également être marquées par des molécules non radioactives telles que la biotine, la digoxégénine, etc. Cependant, l’utilisation de sondes marquées par des isotopes radioactifs est limitée par la courte demi-vie de l’isotope et les aspects économiques et environnementaux de l’élimination des déchets radioactifs.
Les progrès de la technologie des acides nucléiques offrent des alternatives aux sondes marquées par des radioactifs. L’utilisation de marqueurs non radioactifs présente plusieurs avantages tels que la sécurité et une plus grande efficacité de la réaction de marquage. Le marquage des acides nucléiques à la biotine en est un exemple. Ce système exploite l’affinité que la glycoprotéine avidine a pour la biotine. Ces sondes peuvent être préparées à l’avance en vrac et stockées à -20°C pour des utilisations répétées. La digoxigénine est un autre produit chimique dérivé des plantes et utilisé pour le marquage non radioactif des sondes. Un anticorps associé à une enzyme (conjugué antidigoxigénine – phosphatase alcaline) est utilisé pour la détection de la présence de digoxigénine.
Des progrès dans l’imagerie de l’ADN spécifique de la séquence par microscopie à fluorescence ont été réalisés en employant la méthode d’hybridation fluorescente in situ (FISH). Ce type de marquage est particulièrement utile pour l’examen direct de spécimens microbiologiques ou cytologiques au microscope.
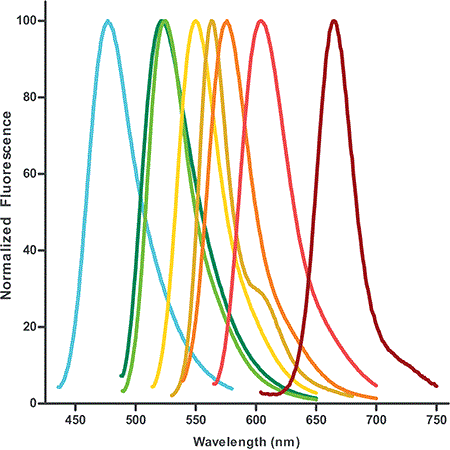
Figure 1 : Profils d’émission de fluorescence des dUTP marqués par fluorescence disponibles. Les dUTP à colorant sont conçus pour être particulièrement performants dans les applications multicolores, telles que l’hybridation in situ et l’analyse des microréseaux.
Qu’est-ce que les sondes ADN ?
Une sonde ADN est un fragment d’ADN qui contient une séquence nucléotidique spécifique du gène ou de la région chromosomique d’intérêt. Les sondes ADN utilisent l’hybridation de l’acide nucléique avec des séquences spécifiquement marquées pour détecter rapidement les séquences complémentaires dans l’échantillon testé. Diverses méthodes de marquage de l’ADN ont été décrites. En résumé, ces méthodes sont utilisées pour générer des sondes marquées en bout ou en continu. La plupart des techniques de marquage à médiation enzymatique sont très dépendantes de l’activité de la polymérase, qui est responsable de l’incorporation des nucléotides marqués. En outre, l’utilisation de Taq ou d’autres ADN polymérases thermostables permet d’effectuer des réactions de marquage à des températures plus élevées via la PCR, ce qui réduit l’incidence des mutations ponctuelles induites par les enzymes pendant la synthèse de la sonde. La PCR est une excellente méthode pour la synthèse des sondes, car elle ne nécessite que de très petites quantités de matériel de matrice. En présence d’amorces nucléotidiques marquées de manière appropriée, les produits de la PCR sont marqués au fur et à mesure qu’ils sont synthétisés. Les amorces elles-mêmes peuvent également être marquées de manière non isotopique pendant leur propre synthèse, ce qui évite d’avoir à inclure des précurseurs nucléotidiques marqués dans le mélange réactionnel. L’amorçage aléatoire est un type d’extension d’amorce dans lequel un mélange de petites séquences d’oligonucléotides, agissant comme des amorces, s’hybrident à une matrice double brin dénaturée par la chaleur. Les amorces recuites deviennent finalement une partie de la sonde elle-même, car le fragment Klenow de l’ADN polymérase I étend les amorces dans la direction 3′ et, ce faisant, incorpore le marqueur. La translation de Nick est l’une des plus anciennes techniques de marquage de sonde. Elle consiste à entailler de façon aléatoire le squelette d’un ADN double brin avec des concentrations diluées de DNase I. À des concentrations extrêmement faibles, cette enzyme entaille une matrice en quatre ou cinq sites, produisant un groupe 3′-OH libre qui peut servir d’amorce à chaque emplacement d’entaille. Ensuite, l’enzyme ADN polymérase I élimine les nucléotides natifs des molécules de la sonde dans le sens 5′→3′ (activité exonucléase) tout en les remplaçant par des précurseurs dNTP marqués en vertu de son activité 5′→3′ polymérase. La traduction Nick est efficace pour les molécules d’ADN linéaires et fermées de manière covalente, et la réaction de marquage est terminée en moins d’une heure.
Enzo propose un système de marquage d’ADN par traduction Nick 2.0 afin de fournir une méthode simple et efficace pour générer de l’ADN marqué. Le kit peut accueillir une large gamme de nucléotides marqués par un fluorophore, par la biotine et par la digoxigénine. En plus du choix du marqueur, la conception du kit permet à l’utilisateur d’optimiser l’incorporation et la taille du produit en ajustant le rapport entre le dUTP marqué et le dTTP. Le mélange enzymatique NT prêt à l’emploi est facile à utiliser et minimise les erreurs de pipetage. Les sondes marquées par la translation nick peuvent être utilisées dans de nombreuses techniques d’hybridation différentes, notamment : l’hybridation in situ (ISH), l’hybridation in situ fluorescente (FISH), le criblage de banques de gènes par hybridation de colonies ou de plaques, l’hybridation par transfert d’ADN ou d’ARN et les études de cinétique de réassociation.
Qu’est-ce que les sondes ARN ?
Les sondes ARN sont des tronçons d’ARN monocaténaires utilisés pour détecter la présence de séquences d’acide nucléique complémentaires (séquences cibles) par hybridation. Les sondes ARN sont généralement marquées, par exemple avec des radio-isotopes, des épitopes, de la biotine ou des fluorophores pour permettre leur détection. Les sondes d’ARN en tant qu’outils d’hybridation restent populaires en raison de plusieurs avantages clés associés à leur utilisation. Ces sondes sont synthétisées par transcription in vitro et peuvent se substituer aux sondes ADN dans presque toutes les applications. Les sondes ARN ou ribosondes à haute activité spécifique peuvent également être synthétisées à partir de modèles d’ADN clonés dans des vecteurs d’expression tels que les systèmes SP 6 et T 7. Les sondes d’ARN sont monocaténaires et offrent plusieurs avantages par rapport aux sondes d’ADN, notamment un meilleur signal ou une meilleure hybridation des blots. Par rapport aux diverses méthodes de synthèse des sondes d’ADN, il n’existe qu’une seule méthode fiable pour marquer les sondes d’ARN, à savoir la transcription in vitro. En raison de la nature intrinsèquement labile de l’ARN et de la susceptibilité à la dégradation par la RNase, les sondes ARN doivent être traitées avec le même soin que toute autre préparation d’ARN.
La transcription in vitro est une méthode fiable et économique pour générer des sondes d’ARN. De grandes quantités de sondes efficacement marquées et de longueur uniforme peuvent être générées par la transcription d’une séquence d’ADN ligaturée à côté d’un promoteur d’ARN. Une excellente stratégie consiste à cloner l’ADN à transcrire entre deux promoteurs d’orientation opposée. Cela permet de transcrire l’un ou l’autre brin de la séquence d’ADN clonée afin de générer de l’ARN sens et antisens pour les études d’hybridation. Une méthode alternative à la génération de sondes ARN marquées en continu par transcription in vitro consiste à marquer l’extrémité 5′ de la molécule. Cette méthode de marquage de l’extrémité 5′ est familièrement connue sous le nom de réaction de kinasing ; elle implique spécifiquement le transfert du γ phosphate de l’ATP à un substrat 5′-OH d’ARN ou d’ADN (réaction directe). La réaction de kinasing en avant est beaucoup plus efficace que la réaction d’échange qui implique la substitution des 5′ phosphates.
Synthèse de sonde par marquage terminal 3′ implique l’addition de nucléotides à l’extrémité 3′ de l’un ou l’autre ADN. Le marquage de l’extrémité 3′ de l’ADN est le plus souvent catalysé par une transférase terminale. Les molécules d’ADN simple et double brin sont marquées par l’addition de dNTP aux extrémités 3′-OH. L’ARN peut également être marqué à l’extrémité 3′ en utilisant l’enzyme poly(A) polymérase. Cette enzyme, qui est naturellement responsable de la polyadénylation nucléaire de nombreux ARN hétéro-nucléaires, catalyse l’incorporation d’Adénosine Mono Phosphate. Le marquage isotopique nécessite des précurseurs d’ATP marqués en α. En plus de son utilité dans les réactions de synthèse de sondes ARN, la poly(A) polymérase peut être utilisée pour polyadényler naturellement les ARNm poly(A) et d’autres ARN afin de soutenir la synthèse d’ADNc médiée par des amorces oligo(dT).
Utilisation des sondes dans les applications de recherche
Dans le Northern blotting, l’ARN étudié est fractionné par électrophorèse sur gel. Les molécules sont ensuite transférées sur une membrane qui est incubée avec la ou les sondes marquées. L’hybridation des séquences complémentaires permet de visualiser la séquence de l’ARN cible. Le transfert de Southern implique le fractionnement et le transfert de l’ADN sur des membranes. Les membranes sont ensuite incubées avec la ou les sondes d’ADN marquées. L’hybridation des séquences complémentaires permet de visualiser la séquence d’ADN cible. D’autres applications impliquant des expériences ISH et FISH permettent la localisation de cibles ARN ou ADN dans les cellules et les tissus. Cette technique utilise des cellules cultivées ou des échantillons de coupes de tissus pour l’hybridation et la détection du gène ou de la séquence cible d’intérêt. Les cellules ou les tissus sont traités de manière à ce que leurs acides nucléiques endogènes soient fixés en place, mais disponibles pour l’hybridation et la détection par des sondes marquées.
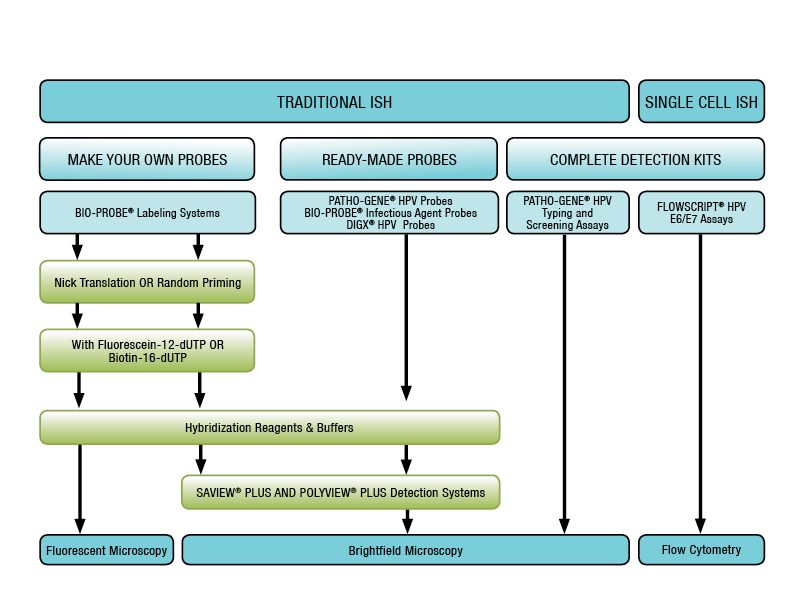
Figure 2 : Enzo Life Sciences offre un ensemble complet de solutions pour l’hybridation in situ, fournissant tout ce dont vous avez besoin pour le marquage, l’hybridation et la détection.
Les avancées dans les technologies d’analyse unicellulaire offrent des perspectives inédites sur l’hétérogénéité phénotypique et fonctionnelle au sein de populations cellulaires apparemment identiques. Les techniques de profilage et de compréhension de l’expression de l’ARN à une résolution unicellulaire ont rapidement progressé ces dernières années.
Enzo Life Sciences est un leader mondial reconnu dans la fourniture de technologies de marquage de l’ADN et de l’ARN avec plusieurs brevets clés dans le développement de la biotine et de la sonde nucléotidique marquée par fluorescence pour les études d’expression génique. Nous offrons une gamme de produits pour les besoins de la recherche en génomique. Pour une méthode simple et efficace de génération d’ADN marqué, veuillez consulter notre kit de marquage d’ADN par traduction de Nick ainsi qu’une liste de nos colorants fluorescents SEEBRIGHT®-dUTP et de notre Allylamine-dUTP. Pour toute question ou préoccupation concernant l’un de nos produits, notre équipe de support technique est là pour vous aider.